Article co-rédigé par Mylène Ruh (GEVES), Thomas BALDWIN (GEVES), Valérie CADOT (GEVES), Denis BEGHIN (GEVES) et Véronique BRAULT (INRAE).
La jaunisse de la betterave étant causée par un complexe de quatre virus présents en mono- ou en co-infection en Europe, il est indispensable de disposer d’un test de détection efficace pour ces quatre virus afin d’évaluer le comportement variétal des betteraves vis-à-vis des différentes jaunisses. Les quatre virus responsables de cette maladie présents sur le territoire français sont :
- BYV (beet yellows virus), le clostérovirus responsable de la jaunisse grave ;
- BChV (beet chlorosis virus) et BMYV (beet mild yellowing virus), les deux polérovirus responsables de la jaunisse modérée ;
- BtMV (beet mosaic virus), le potyvirus responsable de la mosaïque de la betterave
La méthode ELISA employée jusqu’à présent pour déterminer le statut infectieux des plantes est basée sur l’utilisation d’anticorps qui reconnaissent les virus. Cette technique est fonctionnelle, mais ne permet pas de distinguer les deux polérovirus et demande trois analyses distinctes, une par genre viral. Pour répondre à ces deux problématiques, le pôle Détection du BioGEVES, en collaboration avec INRAE de Colmar (équipe de Véronique Brault - projet PNRI ProViBe), a développé une méthode de détection simultanée des quatre virus par RT-qPCR multiplexe dans le cadre du projet PNRI Yellows Resistbeet.
Mise au point de la méthode RT-qPCR multiplexe
La méthode RT-qPCR multiplexe développée repose sur l’amplification simultanée des quatre virus BChV, BMYV, BYV et BtMV. En effet, ce test fait intervenir quatre paires d’amorces permettant d’amplifier spécifiquement le matériel génétique de chacun des quatre virus, et quatre sondes TaqMan marquées par un fluorophore différent, ce qui permet en plus de discriminer de façon spécifique les quatre virus. Ainsi,l’outil développé permet la détection et l’identification des quatre virus responsables de la jaunisse de la betterave en une seule réaction (Figure 1).
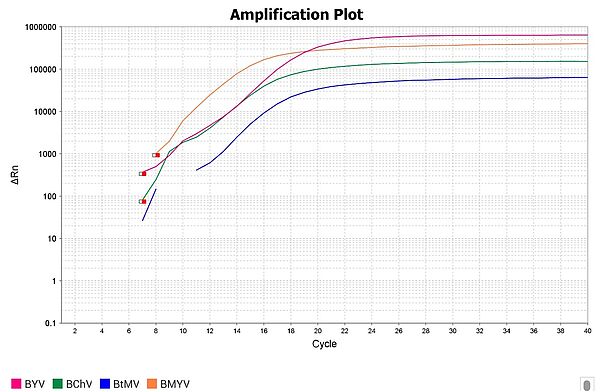
Figure 1 – Exemple d’une analyse de détection par RT-qPCR multiplexe d’un échantillon de feuilles de betterave.Diagramme d’amplification représentant le ΔRn en fonction du nombre de cycles de la RT-qPCR multiplexe, où le ΔRn correspond à l’amplitude du signal de fluorescence généré. Ici, notre échantillon est infecté par les quatre virus et émet donc les quatre signaux de fluorescence représentés par quatre couleurs différentes : en vert pour BChV, en orange pour BMYV, en rose pour BYV et en bleu pour BtMV.
Validation de la méthode RT-qPCR multiplexe
Une fois la méthode de détection mise au point, il est nécessaire de la valider via l’évaluation de différents critères de performance.
La spécificité analytique
L’évaluation de ce critère permet de connaître la capacité de notre méthode à détecter le ou les bioagresseurs cibles (inclusivité) tout en excluant les micro-organismes non-cibles (exclusivité). Pour ce faire, plusieurs échantillons de chacun des quatre virus présents en mono-infection, des échantillons de betterave saine, ainsi que des échantillons contaminés par des virus non-ciblés ont été testés avec notre outil RT-qPCR multiplexe.
Les résultats obtenus ont montré que notre méthode répond à ce critère de spécificité avec une inclusivité et une exclusivité de 100 %. La RT-qPCR multiplexe permet donc de détecter spécifiquement les quatre virus responsables de la jaunisse de la betterave ciblés, et eux seuls.
La sensibilité analytique
Ce critère correspond à la plus petite quantité de bioagresseur cible pouvant être détectée (seuil de détection) par notre méthode. Pour ce faire, nous avons réalisé trois gammes de dilution à partir d’échantillons infectés par chacun des quatre virus et nous avons analysé chaque point de dilution avec la technique ELISA et notre test RT-qPCR multiplexe.
L’évaluation de ce critère a révélé que la méthode RT-qPCR multiplexe est de 100 à 10 000 fois plus sensible que la technique ELISA en fonction du virus ciblé. Notre méthode répond donc à ce critère de sensibilité analytique.
La sensibilité diagnostique
Ce critère permet de s’assurer que notre méthode ne produit pas de faux négatifs. Pour ce faire, nous avons analysé 191 échantillons de feuilles de betteraves infectées par les différents virus et à différents niveaux de contamination avec la technique ELISA et notre outil RT-qPCR multiplexe.
La comparaison des résultats obtenus avec les deux méthodes de détection utilisées a démontré que la RT-qPCR multiplexe ne produit pas de faux négatifs. La sensibilité diagnostique de notre méthode étant de 100 % pour tous les virus ciblés, elle répond à ce critère de sensibilité diagnostique.
La répétabilité et la reproductibilité
L’évaluation de ces critères permet de contrôler la capacité de notre méthode à produire le même résultat dans le même laboratoire par le même opérateur (répétabilité) et le même résultat dans des conditions expérimentales différentes, un autre opérateur ou un autre laboratoire par exemple, (reproductibilité). Pour ce faire, 22 échantillons de feuilles de betterave infectées par les différents virus et à différents niveaux de contamination ont chacun été divisés en quatre sous-échantillons afin d’être analysés au cours de deux tests indépendants par deux opérateurs différents (deux sous-échantillons par opérateur).
Pour tous les virus ciblés, les résultats obtenus ont montré que notre méthode est répétable, avec une répétabilité supérieure à 94 %, et reproductible, avec une reproductibilité supérieure à 95 %. La RT-qPCR multiplexe répond donc à ces deux critères de répétabilité et reproductibilité.
Perspectives
Maintenant que cette méthode de détection et d’identification des quatre virus responsables de la jaunisse de la betterave à moindre coût est validée, elle sera proposée en tant que prestation de service par le GEVES et fera l’objet de différentes valorisations telles qu’une publication dans une revue scientifique et la présentation d’un poster au congrès ICPP (Lyon – août 2023).
De plus, ce procédé d’identification sera utilisé dans le cadre des essais d’évaluation variétale (VATE) orchestrés par le GEVES pour l’inscription de nouvelles variétés de betteraves au Catalogue français, sur proposition du Comité Technique Permanent de la Sélection (CTPS). En effet, le test RT-qPCR multiplexe développé permettra de contrôler en amont les inocula et en aval les inoculations, mais aussi l’éventuelle présence naturelle de virus sur les différents sites utilisés pour l’évaluation variétale. Également, en plus de détecter les virus présents dans les feuilles de betterave, cet outil fournit des données semi-quantitatives et le projet PNRI Yellows Resistbeet étudie actuellement la possibilité d’exploiter ces données pour la caractérisation de la résistance ou la tolérance à la jaunisse des variétés évaluées.
Il est aussi envisageable que cette méthode soit employée dans le cadre de la surveillance épidémiologique pour suivre l'évolution de la jaunisse de la betterave sur le terrain en fonction des années.